Los LOS Neisseria coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from "κορώνη korṓnē" in Greek, which translates as "crown," after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus son un grupo de virus Virus Viruses are infectious, obligate intracellular parasites composed of a nucleic acid core surrounded by a protein capsid. Viruses can be either naked (non-enveloped) or enveloped. The classification of viruses is complex and based on many factors, including type and structure of the nucleoid and capsid, the presence of an envelope, the replication cycle, and the host range. Virology relacionados que contienen ácido ribonucleico (ARN) monocatenario de sentido positivo. El coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from "κορώνη korṓnē" in Greek, which translates as "crown," after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus deriva su nombre de “κορώνα” en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum griego, que se traduce como “corona”, por las pequeñas proteínas en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum forma de mazo visibles como un anillo alrededor de la envoltura viral en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum micrografías electrónicas. Los LOS Neisseria coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from "κορώνη korṓnē" in Greek, which translates as "crown," after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus tienen genomas grandes, son propensos a la mutación y frecuentes eventos de recombinación que han dado lugar a una diversidad de especies. Estas nuevas especies son capaces de adaptarse rápidamente a nuevos huéspedes y entornos ecológicos, lo que las hace HACE Altitude Sickness muy resistentes y difíciles de combatir. Han aparecido nuevas infecciones por coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from "κορώνη korṓnē" in Greek, which translates as "crown," after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus tanto en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum humanos como en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum animales. Se sabe que los LOS Neisseria coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from "κορώνη korṓnē" in Greek, which translates as "crown," after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus son la causa de algunos casos de resfriado común, síndrome respiratorio agudo severo (SARS, por sus siglas en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum inglés), síndrome respiratorio de Oriente Medio (MERS, por sus siglas en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum inglés) y enfermedad por coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from "κορώνη korṓnē" in Greek, which translates as "crown," after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus 2019 ( COVID-19 COVID-19 Coronavirus disease 2019 (COVID-19) is an infectious disease caused by the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) that mainly affects the respiratory system but can also cause damage to other body systems (cardiovascular, gastrointestinal, renal, and central nervous systems). , por sus siglas en EN Erythema nodosum is an immune-mediated panniculitis (inflammation of the subcutaneous fat) caused by a type IV (delayed-type) hypersensitivity reaction. It commonly manifests in young women as tender, erythematous nodules on the shins. Erythema Nodosum inglés).
Last updated: Dec 15, 2025
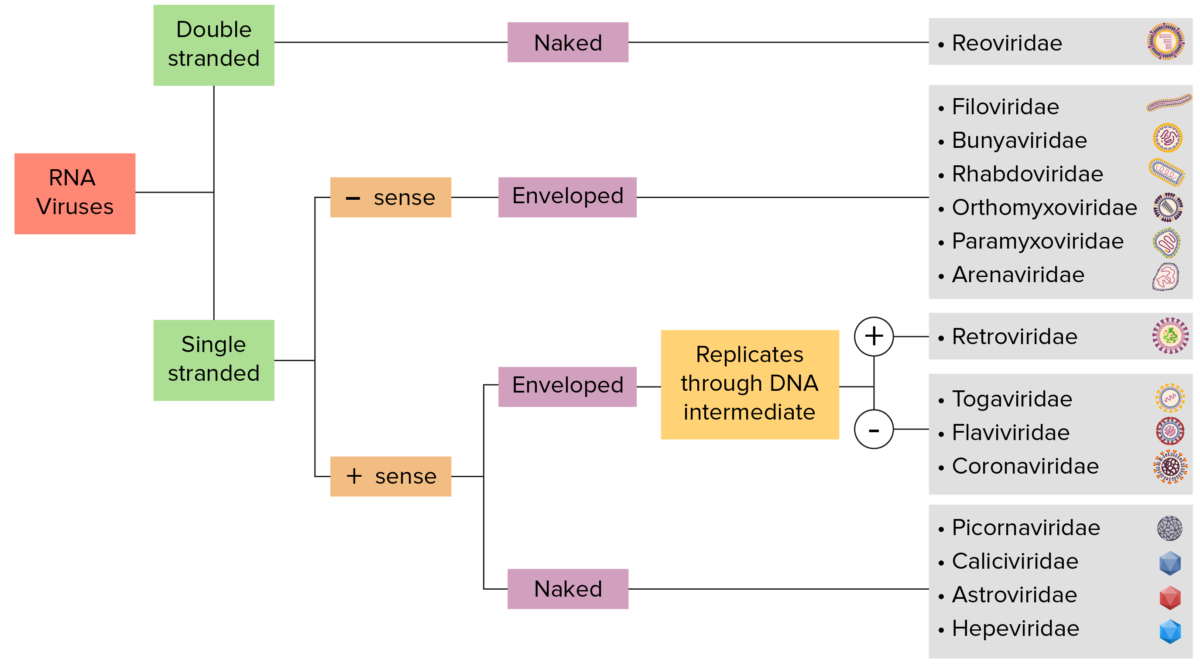
Identificación de virus de ARN:
Los virus se pueden clasificar de muchas formas. La mayoría de los virus, sin embargo, tendrán un genoma formado por ácido desoxirribonucleico (ADN) o ARN. Los virus del genoma de ARN pueden caracterizarse además por ARN monocatenario o bicatenario. Los virus “envueltos” están cubiertos por una fina capa de membrana celular (generalmente extraída de la célula huésped). Si la envoltura está ausente, los virus se denominan virus “desnudos”. Los virus con genomas monocatenarios son virus de “sentido positivo” si el genoma se emplea directamente como ARN mensajero (ARNm), que se traduce en proteínas. Los virus monocatenarios de “sentido negativo” emplean la ARN polimerasa dependiente de ARN, una enzima viral, para transcribir su genoma en ARN mensajero.
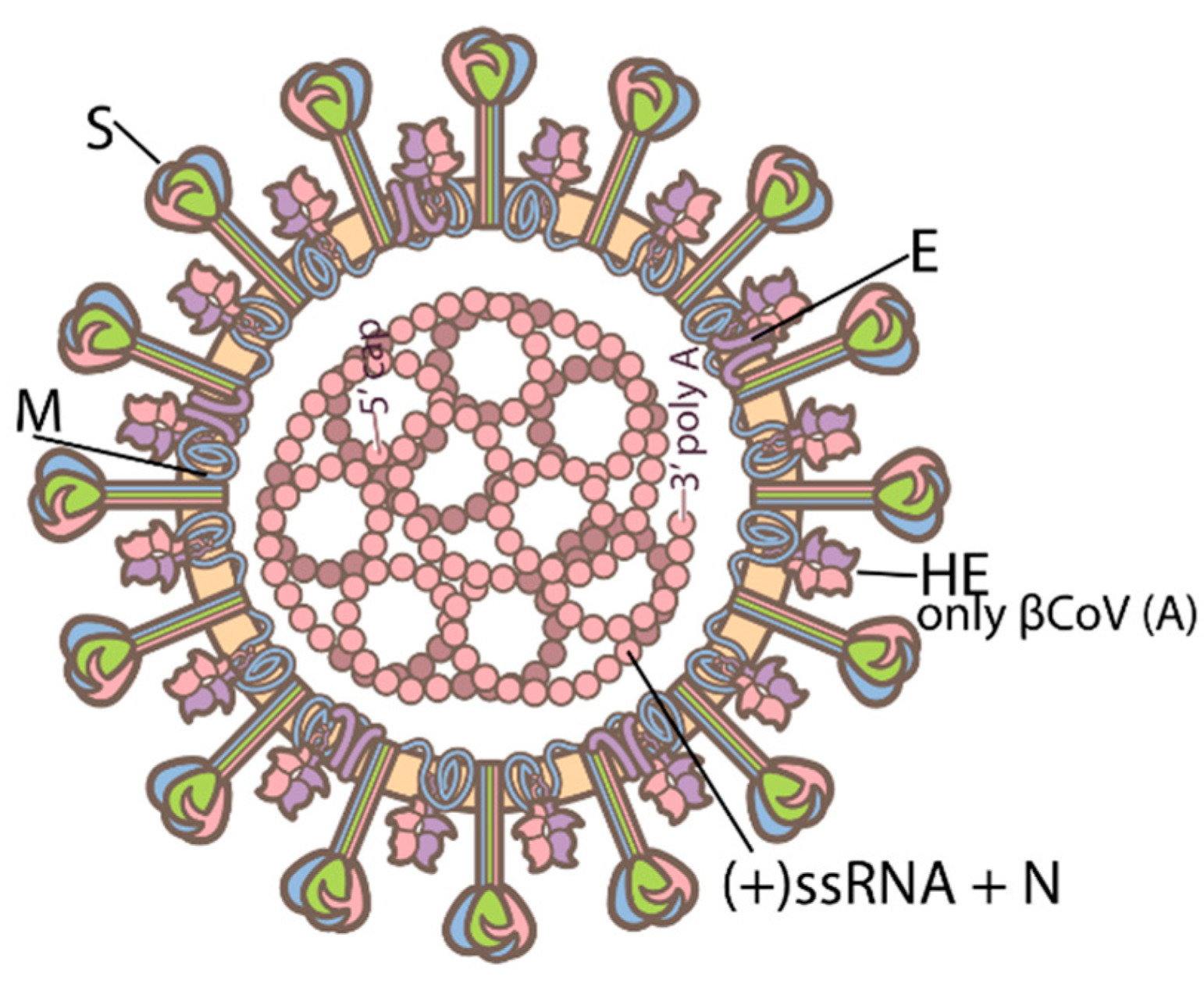
Las proteínas de la envoltura se denominan:
S: espícula
HE: hemaglutinina esterasa
M: membrana
E: envoltura
N: nucleocápside
(+)ssRNA: ARN monocatenario de sentido positivo
La mayoría de coronavirus Coronavirus Coronaviruses are a group of related viruses that contain positive-sense, single-stranded RNA. Coronavirus derives its name from “κορώνη korṓnē” in Greek, which translates as “crown,” after the small club-shaped proteins visible as a ring around the viral envelope in electron micrographs. Coronavirus tienen 4 proteínas estructurales: S, E, M y N.
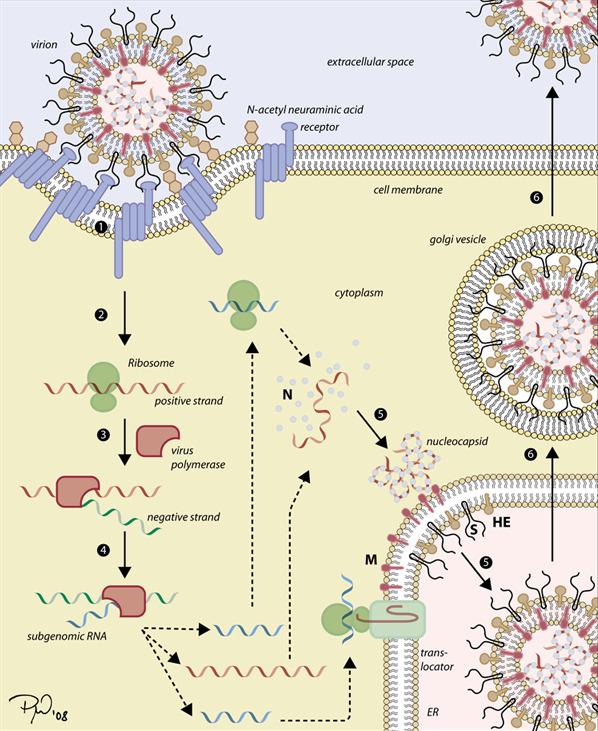
Ciclo de replicación de los coronavirus:
1. Los coronavirus se unen a la superficie de la célula huésped a través de las proteínas S. La entrada viral ocurre por endocitosis mediada por receptores o por fusión de membranas. El virus escapa del entorno acidificado de los endosomas transportándose a los lisosomas.
2. Los coronavirus tienen ARN monocatenario de sentido positivo que puede producir directamente las proteínas y nuevos genomas en el citoplasma.
3. Se produce el ARN molde de cadena de sentido negativo.
4. Las nuevas proteínas virales son traducidas por los ribosomas del huésped.
5. La proteína de la nucleocápside se une al ARN genómico y la proteína M se integra en la membrana del retículo endoplásmico junto con las proteínas S y HE.
6. La nucleocápside ensamblada que contiene el ARN se mueve hacia el retículo endoplásmico para ser envuelta y es liberada por exocitosis.
| MERS-CoV | SARS-CoV SARS-CoV A viral disorder characterized by high fever, dry cough, shortness of breath (dyspnea) or breathing difficulties, and atypical pneumonia. A virus in the genus Coronavirus is the suspected agent. Coronavirus | SARS-CoV-2 | |
|---|---|---|---|
| Fecha del primer caso identificado | Junio 2012 | Noviembre 2002 | Diciembre 2019 |
| Localización del primer caso identificado | Jeddah, Arabia Saudita | Shunde, China | Wuhan, China |
| Edad promedio | 56 años | 44 años | 56 años |
| Proporción de sexos (M:F) | 3,3:1 | 0,8:1 | 1,6:1 |
| MERS (causado por MERS-CoV) | SARS (causado por SARS-CoV SARS-CoV A viral disorder characterized by high fever, dry cough, shortness of breath (dyspnea) or breathing difficulties, and atypical pneumonia. A virus in the genus Coronavirus is the suspected agent. Coronavirus) | COVID-19 COVID-19 Coronavirus disease 2019 (COVID-19) is an infectious disease caused by the severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) that mainly affects the respiratory system but can also cause damage to other body systems (cardiovascular, gastrointestinal, renal, and central nervous systems). (causado por SARS-CoV-2) | |
|---|---|---|---|
| Fiebre | 98% | 99%–100% | 87,9% |
| Tos TOS Thoracic outlet syndrome (TOS) is a broad term used for a spectrum of syndromes related to the general region of the thoracic outlet, which involves the compression or irritation of elements of the brachial plexus, subclavian artery, or subclavian vein. Thoracic Outlet Syndrome seca | 47% | 29%–75% | 67,7% |
| Disnea | 72% | 40%–42% | 18,6% |
| Diarrea | 26% | 20%–25% | 3,7% |
| Dolor Dolor Inflammation de garganta | 21% | 13%–25% | 13,9% |
| Uso del ventilador | 24,5% | 14%–20% | 4,1% |
| Pérdida del gusto/olfato | Rara | Rara | 30–60% |
| Fatiga | Desconocida | 70% | 50–70% |
| Organismo | SARS-CoV-2 | Rinovirus | Coxsackievirus Coxsackievirus Coxsackievirus is a member of a family of viruses called Picornaviridae and the genus Enterovirus. Coxsackieviruses are single-stranded, positive-sense RNA viruses, and are divided into coxsackie group A and B viruses. Both groups of viruses cause upper respiratory infections, rashes, aseptic meningitis, or encephalitis. Coxsackievirus |
|---|---|---|---|
| Características |
|
|
|
| Transmisión |
|
|
|
| Clínica |
|
|
|
| Diagnóstico |
|
Diagnóstico clínico | Diagnóstico clínico |
| Tratamiento |
|
|
|
| Prevención |
|
|
|