A regulação epigenética diz respeito à regulação da expressão génica que não envolve alterações na sequência do DNA ou em qualquer um dos seus produtos da transcrição. As formas mais comuns de regulação epigenética são a metilação do DNA, que suprime a expressão génica, e as modificações nas proteínas das histonas, que afetam a estrutura de "empacotamento" do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure. As modificações epigenéticas são responsáveis pelas condições relacionadas com o imprinting Imprinting The variable phenotypic expression of a gene depending on whether it is of paternal or maternal origin, which is a function of the DNA methylation pattern. Imprinted regions are observed to be more methylated and less transcriptionally active. Epigenetic Regulation, incluindo as síndromes de Prader-Willi e Angelman.
Last updated: Apr 25, 2025
As modificações epigenéticas são modificações no DNA ou no “empacotamento” do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure que afetam a expressão do gene Gene A category of nucleic acid sequences that function as units of heredity and which code for the basic instructions for the development, reproduction, and maintenance of organisms. Basic Terms of Genetics sem alterar o próprio código genético (ou seja, a sequência do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure).
Estas modificações:
A unidade básica de “empacotamento” do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure é o nucleossoma. O DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure envolve 2¼ vezes em torno de um núcleo de 8 proteínas histonas, formando um nucleossoma.
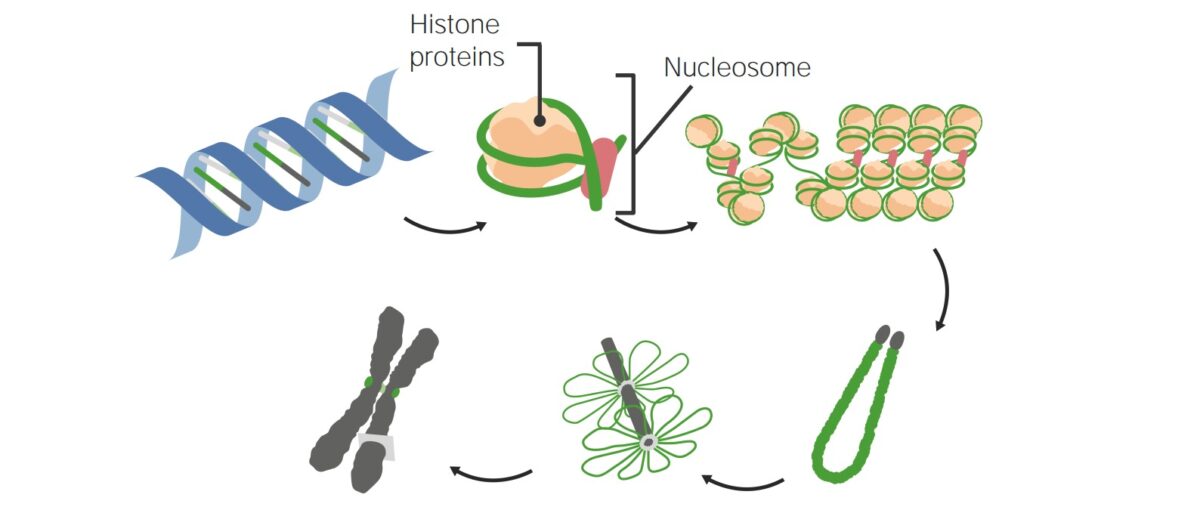
Como o DNA é “empacotado” em nucleossomas e depois em cromossomas
Imagem por Lecturio.As formas mais MAIS Androgen Insensitivity Syndrome importantes de modificação epigenética incluem metilação direta do DNA DNA A deoxyribonucleotide polymer that is the primary genetic material of all cells. Eukaryotic and prokaryotic organisms normally contain DNA in a double-stranded state, yet several important biological processes transiently involve single-stranded regions. DNA, which consists of a polysugar-phosphate backbone possessing projections of purines (adenine and guanine) and pyrimidines (thymine and cytosine), forms a double helix that is held together by hydrogen bonds between these purines and pyrimidines (adenine to thymine and guanine to cytosine). DNA Types and Structure, modificações nas proteínas das histonas e outras modificações na cromatina.
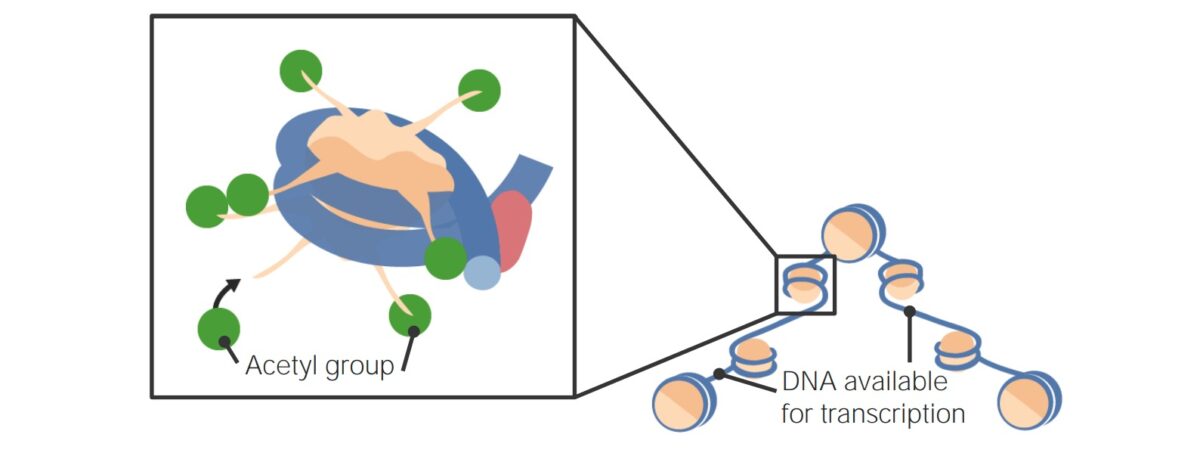
Acetilação de histonas
Imagem por Lecturio.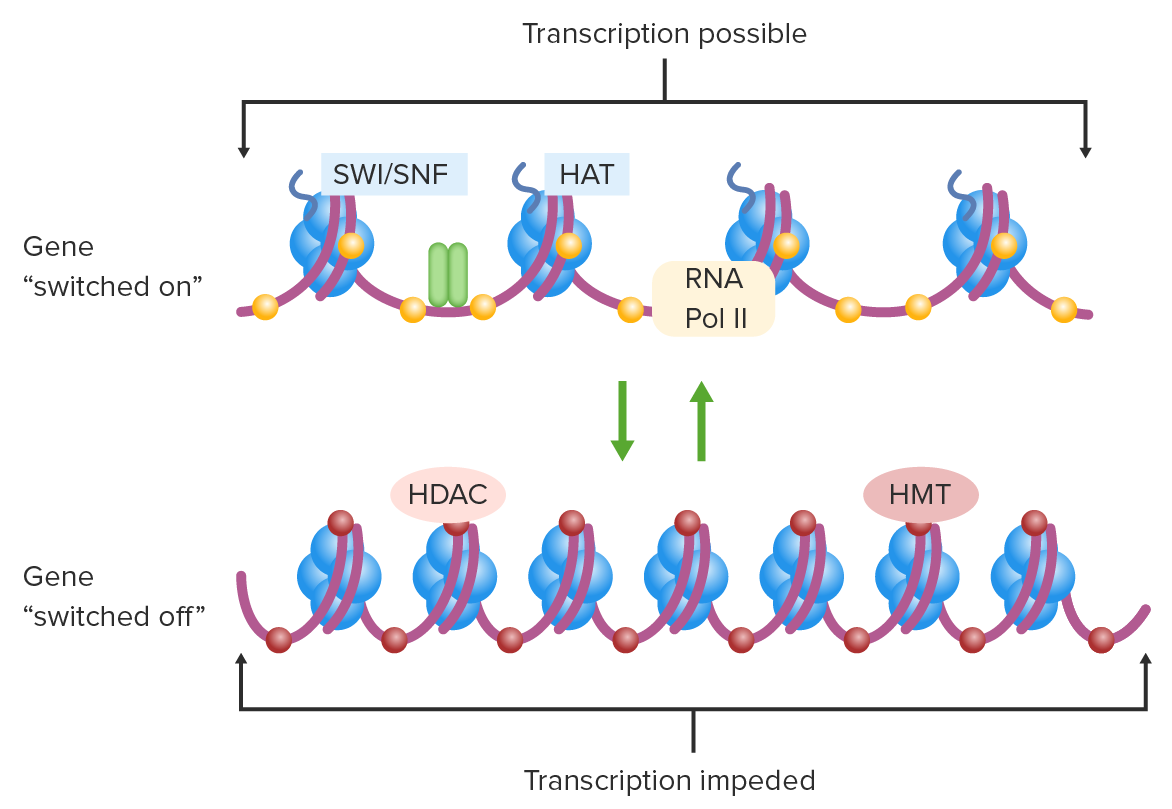
Regulação da transcrição devido a modificações epigenéticas:
Gene ligado: cromatina aberta, citosinas não metiladas, histonas acetiladas.
Gene desligado: cromatina condensada, citosinas metiladas, histonas desacetiladas.
HAT: histona acetiltransferase
HDAC: histona desacetilase
HMT: histona metiltransferase
Pol II: polimerase II
SWI/SNF: switch/ complexo não fermentável de sacarose
A remodelação da cromatina e/ou nucleossomas é outra maneira pela qual a célula pode regular Regular Insulin a expressão génica a nível epigenético. A remodelação da cromatina normalmente requer energia. Os tipos de remodelação da cromatina incluem:
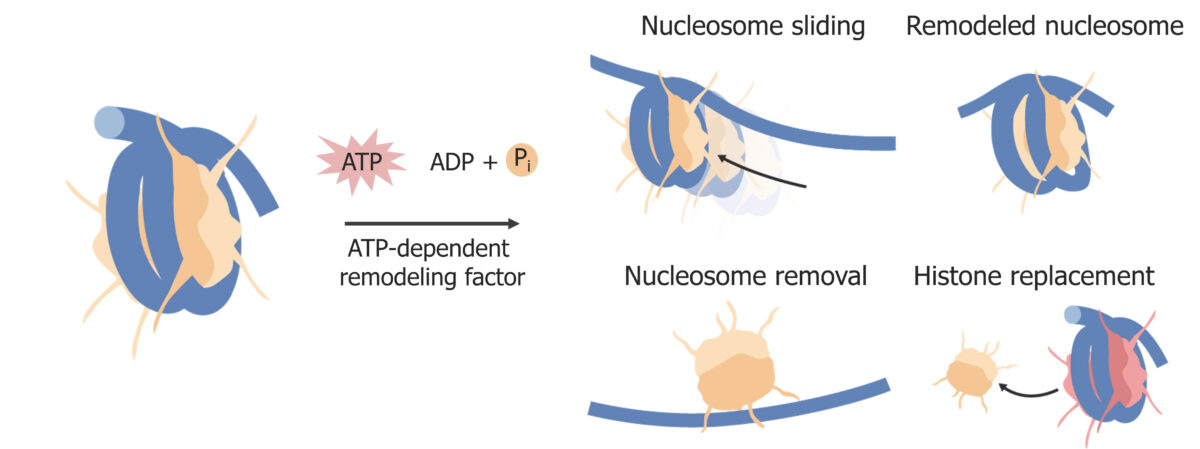
Remodelação de Nucleossomas:
Exemplos de alterações em histonas. Os fatores de remodelação da cromatina também alteram a estrutura da cromatina. A cromatina usa energia do ATP.
O imprinting Imprinting The variable phenotypic expression of a gene depending on whether it is of paternal or maternal origin, which is a function of the DNA methylation pattern. Imprinted regions are observed to be more methylated and less transcriptionally active. Epigenetic Regulation compreende as modificações epigenéticas específicas que ocorrem nos gâmetas sexo-específicos (ou seja, as modificações ocorrem apenas em espermatozoides ou óvulos).
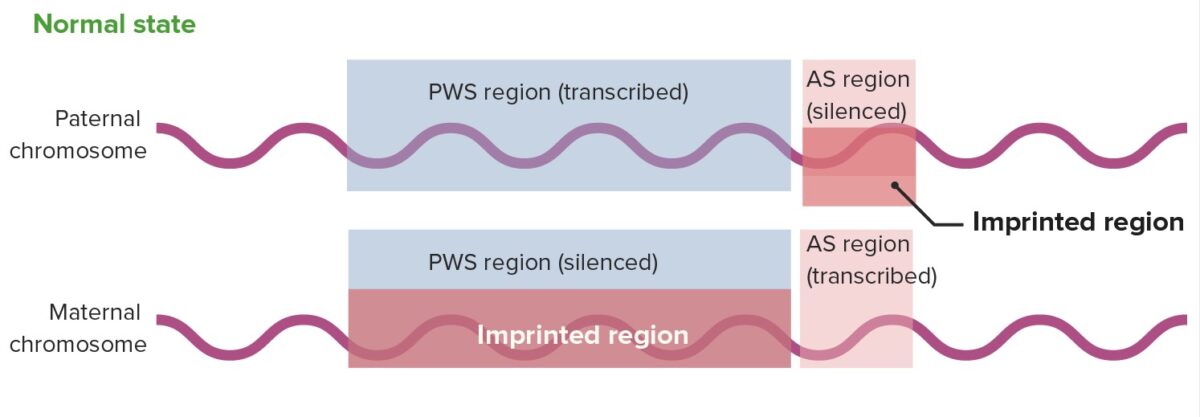
Expressão génica normal na região 15q11-13
AS: Síndrome de Angelman
PWS: Síndrome de Prader-Willi
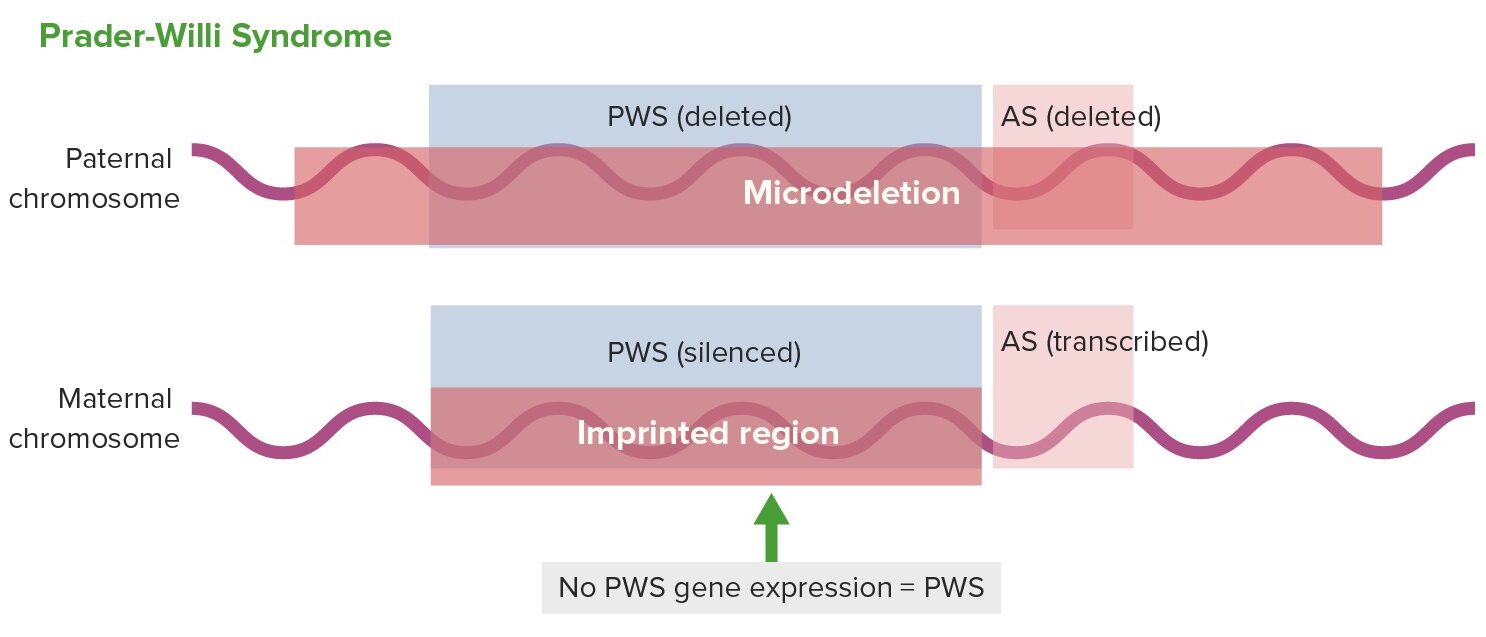
Expressão génica na síndrome de Prader-Willi:
AS: Síndrome de Angelman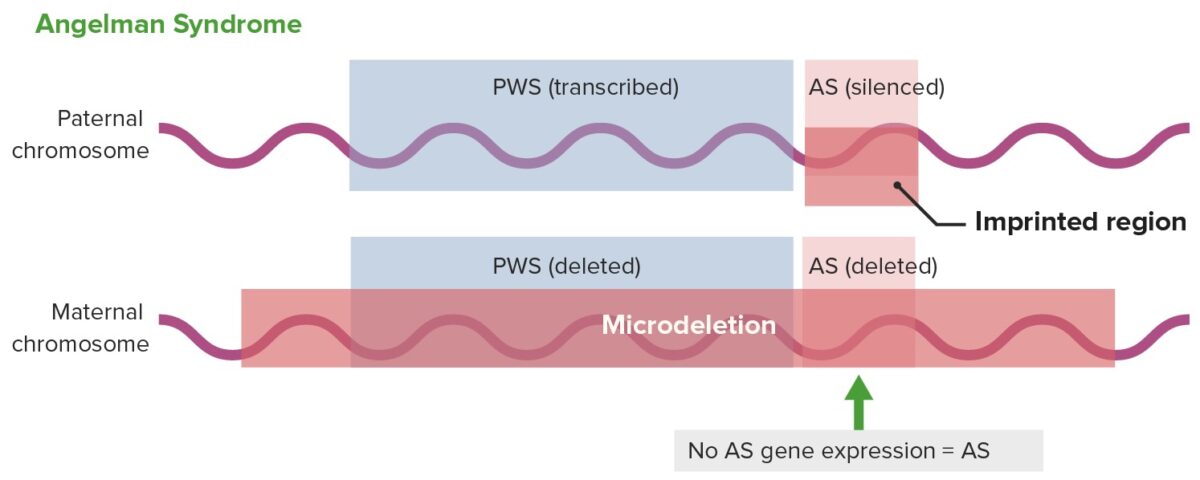
Expressão génica na síndrome de Angelman
AS: Síndrome de Angelman
PWS: Síndrome de Prader-Willi