La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) es una técnica que amplifica exponencialmente fragmentos de ADN para su análisis. El proceso es altamente específico, lo que permite la selección de secuencias genómicas específicas, incluso con cantidades de muestra minúsculas. La PCR cicla múltiples veces a través de 3 fases: desnaturalización del ADN molde, hibridación de un cebador específico con las hebras de ADN individuales y síntesis/elongación de nuevas moléculas de ADN. A partir de ahí, el ADN se puede visualizar con técnicas de análisis como la electroforesis en gel. La velocidad, el bajo costo, la facilidad de uso y la alta sensibilidad y especificidad hacen que esta técnica sea muy útil para las ciencias básicas y biomédicas, y se ha vuelto fundamental en muchas aplicaciones, incluido el análisis forense, el diagnóstico de enfermedades infecciosas y el diagnóstico y tamizaje de anomalías genéticas.
Última actualización: Jun 21, 2025
La reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) es una técnica que amplifica secuencias de ADN específicas in vitro y de manera exponencial y se utiliza para el diagnóstico, la identificación de virus y el análisis.
Para amplificar el ADN o el ARN, las siguientes etapas se repiten en ciclos (a menudo hasta 30–40 veces):
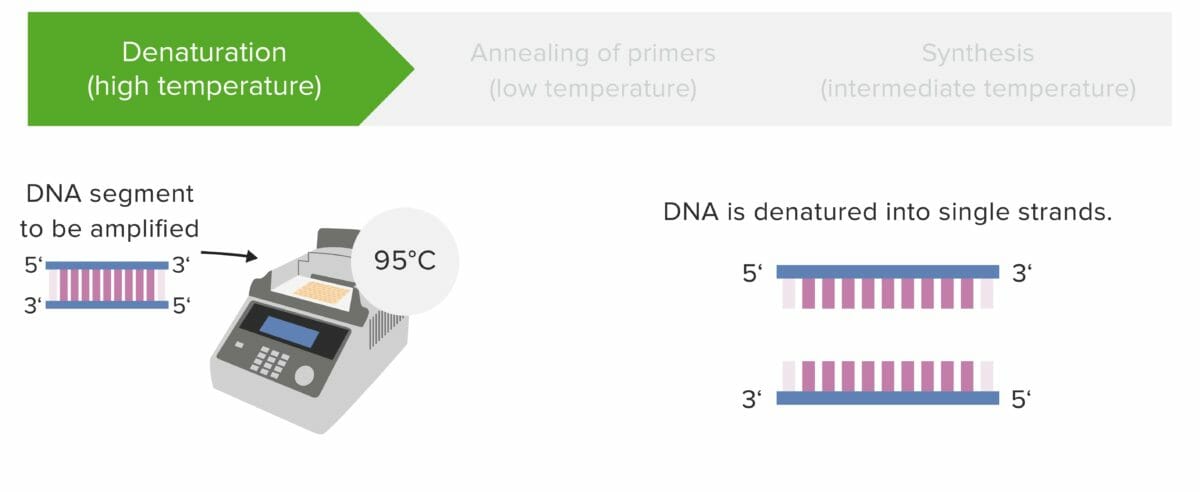
Etapa 1: Desnaturalización
El ADN se desnaturaliza a altas temperaturas, provocando la separación en hebras únicas.
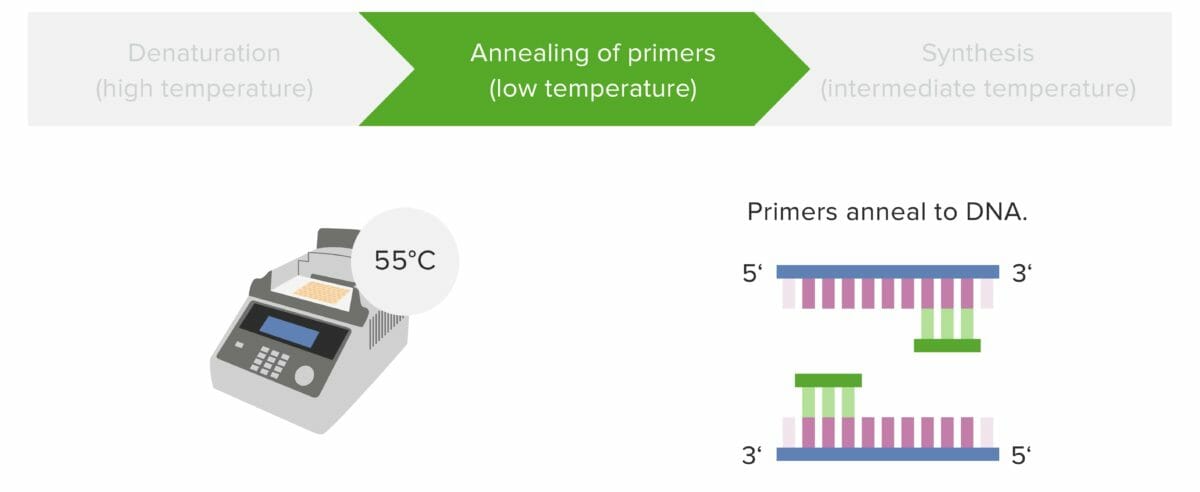
Etapa 2: Hibridación de los cebadores
Los cebadores se hibridan con el extremo 3′ de las hebras de ADN molde al enfriar la muestra.
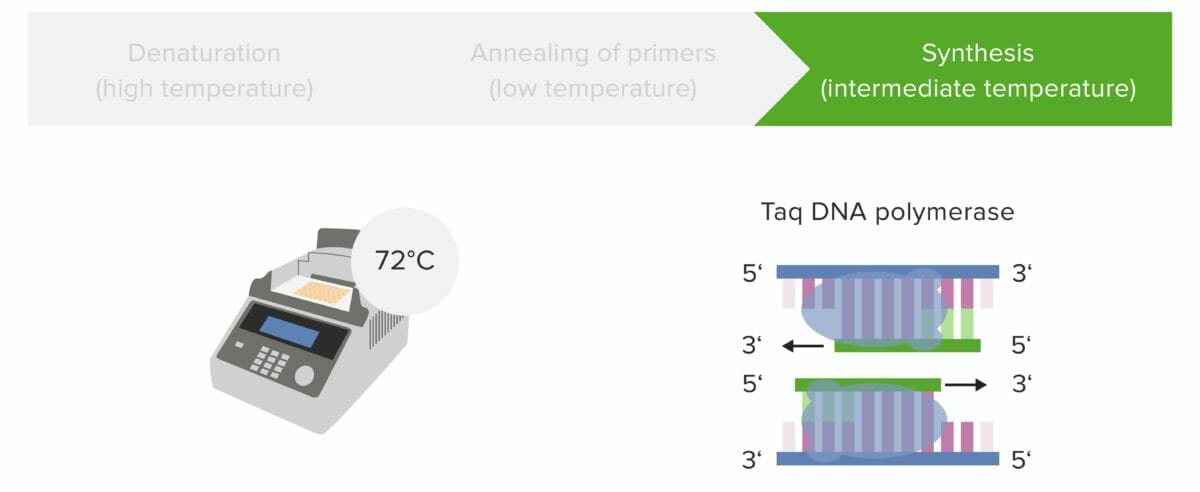
Paso 3: Síntesis/elongación
La elongación del ADN es catalizada por una ADN polimerasa termoestable, que agrega nucleótidos libres disponibles complementarios a la hebra del ADN molde, lo que da como resultado una molécula de ADN recién sintetizada. El ADN se calienta a 72°C, la temperatura adecuada para que la ADN polimerasa Taq pueda elongar los cebadores.
Hay muchas variaciones de la técnica básica de PCR. Algunos de los más comunes incluyen:
La PCR ha mejorado significativamente el potencial diagnóstico de enfermedades infecciosas.